Los científicos y los investigadores de la salud, que antes estaban limitados por el número de muestras que podrían estudiarse en un laboratorio húmedo o la calidad de los microscopios para mirar a las células, están aprovechando poderosas herramientas computacionales para extraer información de un tesoro cada vez mayor de datos biológicos.
Detrás de esta revolución de la biología digital hay una combinación de sistemas de computación de alto rendimiento y frameworks de software específicos de dominio.
El día de hoy, se presentaron dos supercomputadoras que aparecen en el ranking TOP500 de los sistemas más potentes: Cambridge-1 de NVIDIA, centrada en la atención médica, y BioHive-1, de la compañía de biotecnología Recursion, se basan en la arquitectura de referencia NVIDIA DGX SuperPOD.
Y las instituciones de investigación médica, las compañías farmacéuticas y las nuevas empresas de biotecnología de todo el mundo utilizan NVIDIA Clara Parabricks, un conjunto de bibliotecas genómicas y aplicaciones de referencia, para impulsar los workflows de secuenciación de próxima generación.
Mingma Biotechnology, con sede en Shanghái, se convirtió este mes en el primer laboratorio de investigación en China en lanzar Clara Parabricks Pipelines para apoyar su trabajo en medicina de precisión. Esto se suma a las iniciativas de genómica a gran escala que se implementaron en Tailandia y Japón este año. Y la startup de terapia de genes Greffex adoptó recientemente Parabricks Pipelines para acelerar su proyecto de desarrollar una vacuna universal contra la influenza.
Identificar Conocimientos Genómicos para Estudios de Población
Parabricks Pipelines acelera los proyectos basados en ADN y ARN hasta 50 veces con las GPU de NVIDIA, lo que permite a los científicos extraer tanta información útil como sea posible de los cientos de terabytes de datos de instrumentos generados diariamente. Esta aceleración es especialmente poderosa para las instituciones de salud pública y los laboratorios de investigación que ejecutan estudios de población con decenas de miles de genomas para ser analizados.
Mingma Biotechnology adoptó Parabricks Pipelines y las GPU NVIDIA T4 Tensor Core para acelerar su trabajo en secuenciación y análisis de datos multiómicos. La compañía proporciona a las instituciones médicas, compañías farmacéuticas e investigadores información genómica para la investigación de enfermedades y el desarrollo de fármacos.
En el Biobanco Nacional de Tailandia, un sistema NVIDIA DGX A100 impulsa Genomics Thailand, una iniciativa para introducir la medicina genómica como un servicio de salud común en el país. La institución de investigación está utilizando Parabricks Pipelines para analizar las variaciones genéticas de los datos de secuenciación del genoma completo de 50,000 voluntarios tailandeses.
El uso en conjunto del sistema DGX con Parabricks Pipelines redujo el tiempo de procesamiento de datos del genoma completo del proyecto en cuatro meses. Las ideas de este trabajo ayudarán a los investigadores a analizar mejor la variación genética específica de la población tailandesa.
Y en Japón, el Centro del Genoma Humano de la Universidad de Tokio lanzó recientemente SHIROKANE, la supercomputadora más rápida del país para las ciencias de la vida. El sistema con motor DGX A100 está ejecutando Parabricks Pipelines para secuenciar genomas completos de 92,000 pacientes, lo que crea una base de datos que es fundamental para los esfuerzos de medicina de precisión para el cáncer y las enfermedades intratables.
Potenciar la Secuenciación Clínica y el Descubrimiento de Fármacos
El conjunto de herramientas genéticas de Parabricks Pipelines se puede configurar para satisfacer las necesidades específicas de cada laboratorio. Los investigadores ejecutan cargas de trabajo de Parabricks Pipelines en sistemas con las GPU de NVIDIA, que van desde workstations de desktops hasta clouds acelerados por GPU y algunas de las supercomputadoras más rápidas del mundo.
A las pocas semanas de comenzar con una workstation de ciencia de datos con GPU NVIDIA RTX, Greffex, con sede en Houston, está utilizando Parabricks Pipelines y NVIDIA Clara Discovery para avanzar en sus esfuerzos por desarrollar una vacuna universal contra la influenza.
La startup utiliza una combinación de secuenciación genómica, herramientas de dinámica molecular e investigación de laboratorio húmedo para estudiar cómo evolucionan las cepas de influenza con el tiempo y cómo estas mutaciones afectan la eficacia de la vacuna.
Para monitorear los cambios en la gripe, Greffex recopila decenas de miles de genomas de la gripe de todo el mundo y ejecuta enormes alineaciones de secuencias en las GPU NVIDIA RTX 8000 para identificar dónde está cambiando el código genético del virus. La ejecución de cargas de trabajo genómicas en GPU está ahorrando a la empresa hasta 13 horas por muestra, al mismo tiempo que permite a su equipo volver a ejecutar muestras con diferentes parámetros para ajustar los resultados de alineación.
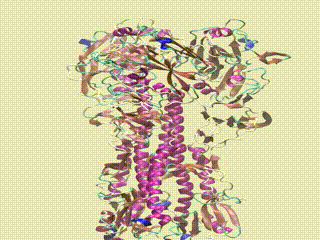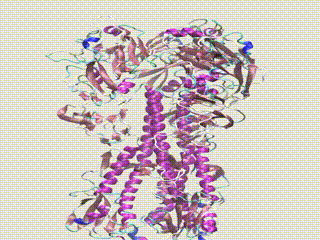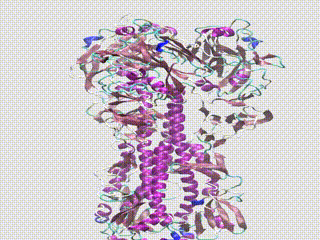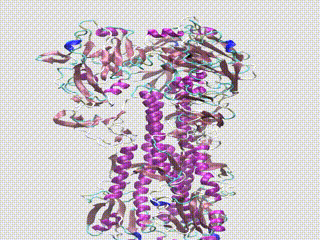
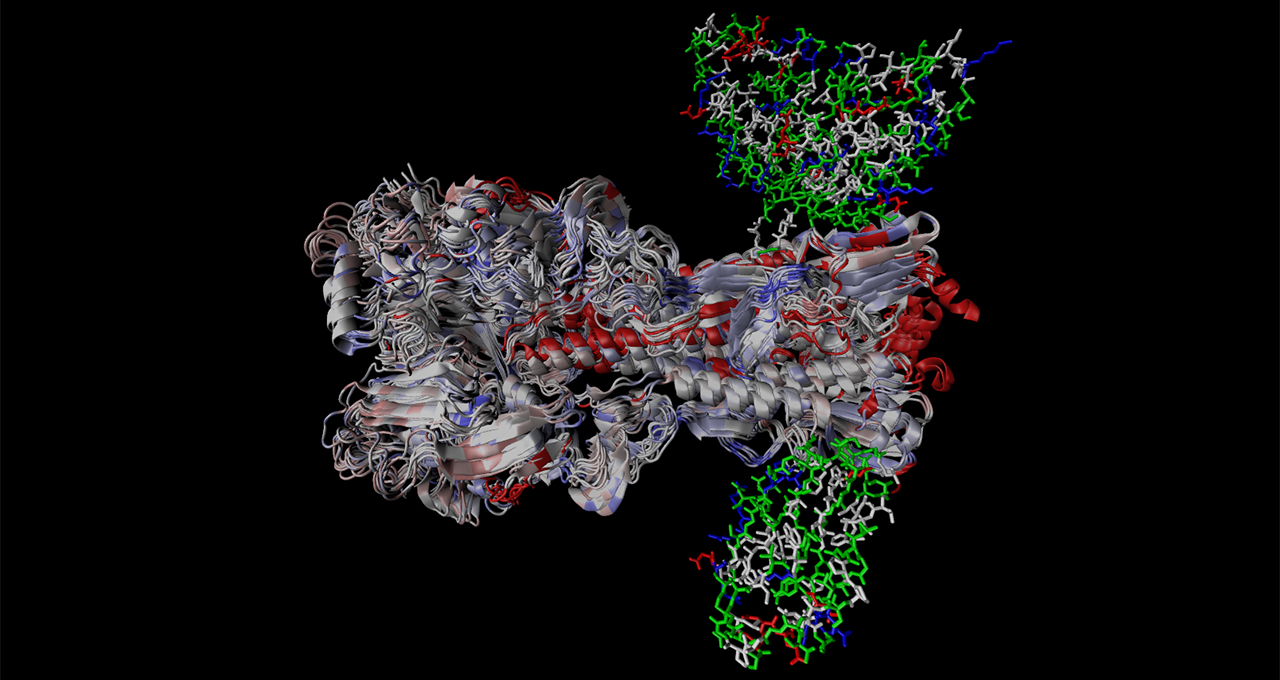
Una vez que se identifican las variantes genéticas, los científicos de Greffex utilizan la dinámica molecular para visualizar cómo estos cambios genéticos alteran la forma física del virus de la influenza. Están atentos a las mutaciones divergentes, donde el virus de la influenza puede transformarse a una forma que no se une tan bien con los anticuerpos impulsados por la vacuna.
“Es un proceso muy largo y costoso para optimizar la estructura de la proteína para una vacuna que se une no solo con la cepa actual de la gripe, sino con un montón de otras cepas”, dijo Daniel Preston, científico de bioinformática de Greffex. “Con los métodos computacionales, podemos tener una idea de lo que probablemente funcionará antes de realizar las pruebas en laboratorios del mundo real. Es como usar un bisturí en lugar de usar un martillo”.
Información sobre NVIDIA Clara Parabricks
NVIDIA Clara Parabricks aporta aceleraciones de GPU al kit de herramientas de análisis del genoma estándar de la industria del Broad Institute, así como herramientas populares como el llamador genético DeepVariant de Google. Al ejecutarse en una GPU NVIDIA A100 Tensor Core, Parabricks acelera los tiempos de análisis secundario de todo un genoma humano a 23 minutos para la llamada de variante de línea germinal de ADN, en comparación con más de 20 horas en un sistema de CPU.
Comenzando con las lecturas de secuenciación de ADN, Clara Parabricks Pipelines puede alinear, clasificar, filtrar y llamar a variantes para la detección de variantes somáticas y de línea germinal, así como admitir aplicaciones basadas en ARN. Las variantes de la línea germinal son aquellas heredadas a través de la ascendencia de un individuo, mientras que las mutaciones somáticas ocurren en las células humanas a lo largo de la vida de una persona y pueden provocar cáncer.
La versión 3.6 de Parabricks Pipelines, que se publicará el próximo mes, ofrecerá más herramientas para la llamada de variantes somáticas , que brinda a los investigadores información para la oncología de precisión, y la llamada de variante de línea germinal de novo, que informa la investigación de enfermedades complejas como el autismo.
La variante de línea germinal de novo se desarrolló en colaboración con investigadores de la Facultad de Medicina de la Universidad de Washington. La técnica reduce el tiempo de ejecución a menos de una hora para analizar los datos del genoma e identificar nuevas variantes dentro de una ascendencia familiar o un trío padres-hijo.
Comienza a utilizar NVIDIA Clara Parabricks Pipelines para el análisis acelerado del genoma en NGC o en AWS Marketplace.
La imagen principal muestra una alineación estructural de 17 proteínas H1 hemaglutinina que abarcan 102 años con anticuerpos unidos simulados. Los segmentos coloreados corresponden a diferentes tipos de mutaciones en las proteínas. Imagen cortesía de Greffex, utilizando modelos del Banco de Datos de Proteínas RCSB.